Componentes Principais
# para exemplificar a utilização da análise de componentes principais ou principal component analysis (PCA), vamos considerar as médias das medidas de comprimento dos dígitos de 15 animais nelore, 15 curraleiros, 15 pantaneiros e 12 búfalos, contidas no quadro abaixo (dados parciais da dissertação de Silva 2012)
| A | B | C | D | E | |
| Búfalo | 0.34 | 0.72 | 0.61 | 0.63 | 5.39 |
| Curraleiro | 0.60 | 0.46 | 0.26 | 0.49 | 4.54 |
| Nelore | 0.86 | 0.64 | 0.33 | 0.53 | 4.95 |
| Pantaneiro | 0.59 | 0.52 | 0.30 | 0.53 | 4.56 |
# baixe os dados via web
dados<-read.table("https://19e31fa08e.cbaul-cdnwnd.com/fd58c287e8dcea635dc0088984f5a026/200000016-b6ee0b765f/digitos.txt",header=TRUE, sep="", dec=".")
# após entrar com a tabela acima em um objeto (data.frame) denominado de "dados", obtemos os componentes principais utilizando a função "prcomp"
cp=prcomp(~A+B+C+D+E, scale = TRUE, data=dados)
# a análise é gravada em um objeto aqui denominado "cp"
# na função deve-se acrescentar o nome das variáveis, o argumento scale (opcional mas recomendado) refere-se a padronização dos dados (TRUE=padroniza os dados) e o argumento data indica o nome do objeto onde estão contidas as variáveis
# a importância de cada variável para a variância obtida em cada componente é tanto maior quanto maior for o valor (em módulo) associado a variável
# observe abaixo que para o primeiro componente todas as variáveis contribuem para a variância e tem importância no estudo (apesar de pequena superioridade da variável C)
# para o segundo componente a variável de maior importância é a A
Standard deviations:
[1] 2.033349e+00 9.077014e-01 2.038829e-01 5.646475e-16
Rotation:
PC1 PC2 PC3 PC4
A -0.3204996 -0.83524822 0.1099610 0.38065822
B 0.4454724 -0.46343394 0.2486452 -0.37843193
C 0.4899380 0.08733382 -0.1748664 0.80383126
D 0.4865881 0.05482483 0.6689900 0.03392931
E 0.4711975 -0.27741022 -0.6692969 -0.25415095
# pode-se também observar a importância de cada variável pela correlação entre a matriz de correlação dos dados com a matriz de componentes (chega-se a mesma conclusão acima)
cor(cor(dados), cp[[2]])
PC1 PC2 PC3 PC4
A -0.9777456 -0.9264544 0.03186677 0.0943770
B 0.9885728 0.7328249 -0.10800235 -0.3879260
C 0.9997947 0.8378016 -0.08120713 -0.2516533
D 0.9996691 0.8367275 -0.05927786 -0.2586718
E 0.9961929 0.7762294 -0.12236727 -0.3297475
# uma outra informação importânte é o quanto da variância é acumulada com os dois primeiros componentes
summary(cp)
Importance of components:
PC1 PC2 PC3 PC4
Standard deviation 2.0333 0.9077 0.20388 5.646e-16
Proportion of Variance 0.8269 0.1648 0.00831 0.000e+00
Cumulative Proportion 0.8269 0.9917 1.00000 1.000e+00
# a variância acumulada deve ser de no mínimo de 70% para a análise ser considerada acurada, sendo que neste caso atinge 99%
# com o gráfico dos dois primeiros componentes pode-se avaliar a similaridade entre os grupos e as correlações entre as variáveis
biplot(cp)
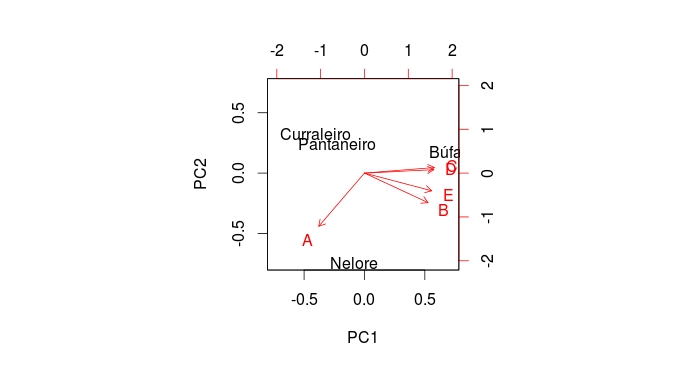
# fazendo alguns ajustes no gráfico
biplot(cp, xlim=c(-0.9,0.9), ylim=c(-0.8,0.8), ylab="Componente 2 (16%)", xlab="Componente 1 (83%)", col=1)
abline(h=0, v=0, lty=2, col=2)
points(x=0,y=0,pch=1,cex=30, col="dark blue")
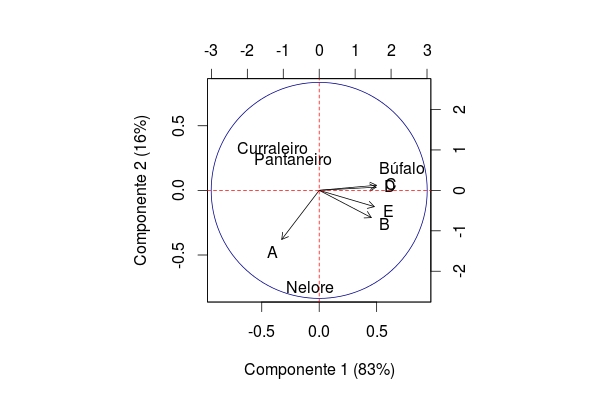
# observa-se que os curraleiros e os pantaneiros são mais similares (mais próximos no gráfico) e os nelores e búfalos mais distantes são menos similares
# as variáveis C e D são correlacionadas positivamente (com o angulo aproximando de zero a correlação aproxima de +1)
# as variávies A e B tem correlação próximo a zero (com angulo aproximando a 45 graus a correlação aproxima a 0)
# as variáveis A e C tem correlação negativa (quando angulo aproximando a 180 graus a correlação aproxima de -1)
# veja as correlações de pearson e compare com o gráfico de componentes principais
require(ds)
dscor(dados)
pairs correlation p-value 1 A and B -0.2702 0.7298 2 A and C -0.7101 0.2899 3 A and D -0.6795 0.3205 4 A and E -0.4365 0.5635 5 B and C 0.8672 0.1328 6 B and D 0.8822 0.1178 7 B and E 0.9669 0.0331 8 C and D 0.9847 0.0153 9 C and E 0.9394 0.0606 10 D and E 0.9168 0.0832
# por fim pode-se obter gráficos com maior controle
c=cp$x
c=as.data.frame(c)
attach(c)
x1=c$PC1# primeiro componente
x2=c$PC2 # segundo componente
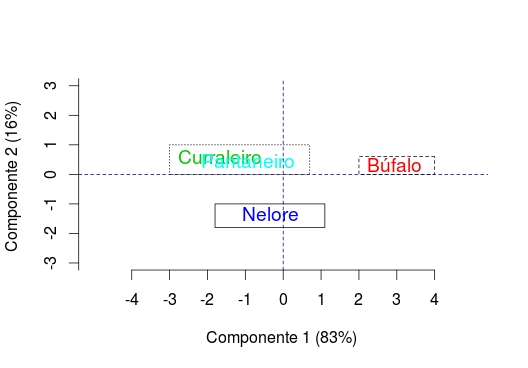
# obter o gráfico para observar a similaridade entre os grupos
plot(x2~x1, ylab= "Componente 2 (16%) ", xlab= "Componente 1 (83%) ", cex=0, xlim=c(-5,5), ylim=c(-3,3), bty="n", axes=FALSE)
axis(1,c(-4,-3,-2,-1,0,1,2,3,4))
axis(2,c(-4,-3,-2,-1,0,1,2,3,4))
abline(v=0,h=0, lty=2, col="dark blue")
rect(4,0.6,2,0, lty=2)
rect(-1.8,-1.8,1.1,-1, lty=1)
rect(-3,1,0.7,0, lty=3)
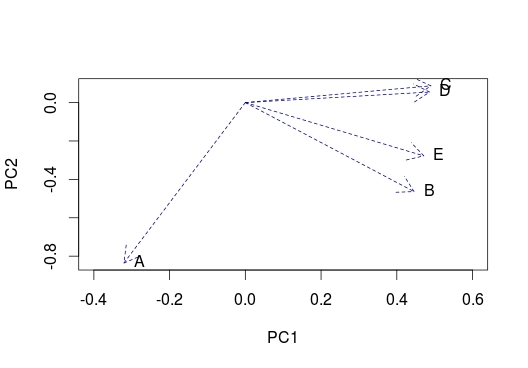
# gráfico para observar as correlações
cpr=as.data.frame(cp$rotation)
y1=cpr$PC1
y2=cpr$PC2
plot(y1,y2, cex=0, xlim=c(-0.4,0.6), xlab="PC1", ylab="PC2")
text(y1,y2, label=rownames(cpr), pos=4)
arrows(0,0,y1,y2, lty=2, col="dark blue")
Referências
Silva, L.H. Morfometria radiográfica e tomográfica de dígitos de bovinos e bubalinos. Dissertação. Universidade Federal de Goiás. Curso de Pós Graduação em Ciência Animal. 63p.
